Aulas Teórico-Práticas de Bioquímica
|
| Bases de Dados |
| Alinhamento de sequências |
| OMIM |
| Visualização de proteínas 2 |
| Árvores Filogenéticas |
Docking de proteínas
Utilização do AutoDockTools (ADT) e AutoDock Vina
1. Criar uma pasta de projecto onde vai realizar todos os ensaios de docking. Pode ser em qualquer local no computador ou mesmo numa pen. Nesta pasta vai gravar todos os ficheiros que vai criar durante este módulo. Neste exemplo tudo é feito numa pen. Mas aconselho a gravar numa pasta do tipo C:/docking ou D:/docking para simplificar os passos seguintes.
2. Fazer o download do programa ADT (seta vermelha): http://mgltools.scripps.edu/downloads.
Instalar como qualquer programa, dizer sim a tudo. Este programa é de acesso livre para instituições académicas e pode ser utilizado livremente.
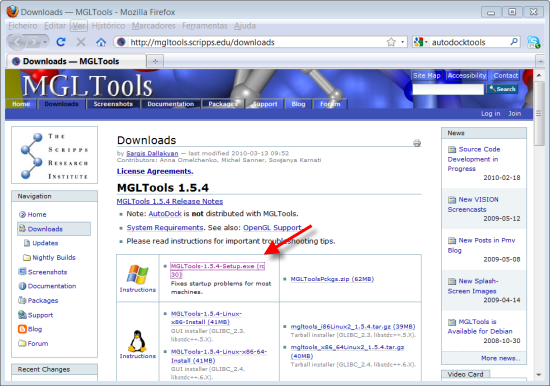
3. Deverá aparecer um icon com o nome do programa. Fazer duplo click no icon para abrir AutodockTools (ADT).

Com o ADT abrem duas janelas mas aquela onde vamos trabalhar é a que está apresentada acima (nunca fechar a segunda janela ou o trabalho desaparece!). O ADT permite preparar as proteínas e ligandos para docking bem como visualizar os resultados no fim do docking. Faz tudo menos o docking propriamente dito que é realizado pelo AutoDock Vina.
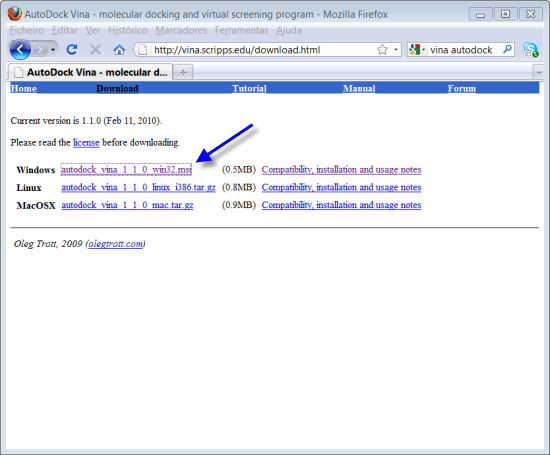
4. Fazer o download do programa Vina (seta azul): http://vina.scripps.edu/download.html.
Instalar como qualquer programa, dizer sim a tudo.
Atenção! Ao instalar o programa escolher para pasta de instalação a pasta de projecto que criou no primeiro passo. Não é essencial mas facilita o trabalho de docking mais à frente. Este programa também é de acesso livre para instituições académicas e pode ser utilizado livremente.
5. No caso deste tutorial a pasta a utilizar chama-se "docking" e nesta fase deverá apresentar os seguintes ficheiros.

O programa que vamos utilizar é o vina.exe.
6. Agora que está tudo pronto vamos começar o trabalho propriamente dito que está dividido em 3 partes:
(1) preparação da proteína e ligandos(s) - ADT
(2) realização do docking - Vina
(3) analizar os resultados do docking - ADT
(1) preparação da proteína e ligandos(s) - ADT
7. Vamos começar por fazer o donwload da estrutura 3D da proteína que vamos utilizar como exemplo. Neste caso é a estrutura cristalografada do domínio cinase da proteína c-Abl. Esta proteína alterada aparece em várias situações de cancro especialmente em casos de leucemias, e resulta da fusão de dois genes sendo expressa numa forma alterada da proteína que está constantemente activa. A estrutura encontra-se depositada no Protein Data Bank (PDB) complexada com um conhecido inibidor (ligando), o imatinib (STI-571) utilizado clinicamente em terapia antitumoral. Para fazer o download da estrutura aceder ao site:
http://www.rcsb.org/pdb/home/home.do

8. Pesquisar o código da estrutura que vamos utilizar : 1IEP (seta amarela). Uma vez na página da estrutura clicar na secção "Download Files" e escolher "PDB file (Text)" (setas verdes).

Como pode observar na página, a base de estruturas proteícas Protein Data Bank tem neste momento cerca de 65000 estruturas.
9.Vai aparecer a seguinte janela. Escolher "Gravar ficheiro" e gravar o ficheiro na pasta do projecto.
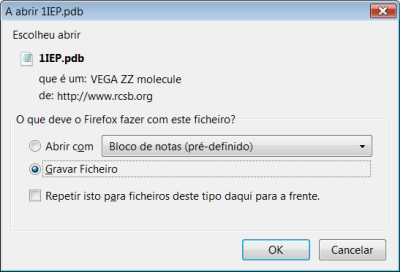
10. Abrir o ficheiro 1IEP.pdb com o wordpad (não com o notepad nem com o Window Word pois aparece tudo desformatado). Antes de começar o trabalho temos que editar o ficheiro 1IEP.pdb da proteína que vamos utilizar. No ficheiro apagar todas as linhas correspondentes aos HETATM (ligandos e águas), a linha TER e as linhas CONNECT. A parte final do ficheiro deverá ficar como o apresentado na figura.
O objectivo deste passo é remover da estrutura da proteína as águas que estão à volta da estrutura e o ligando imatinib (STI-571) pois este iria interferir com o processo de docking que vamos realizar.
Os ficheiros pdb contêm a informação das coordenadas espaciais (x,y,z) de todos os átomos da proteína, nesta proteína 4459 átomos e 498 aminoácidos.
11. Gravar o ficheiro com o nome 1IEP-rec.pdb na pasta do projecto. Não esquecer a terminação pdb. Fechar o ficheiro.
12. Voltar a abrir o ficheiro 1IEP.pde com o wordpad. Agora apagar tudo menos as linhas que começam com HETATOM e que tenham a expressão STI e A (seta roxa). A melhor forma de fazer isto é seleccionar estes linhas escolher CUT, apagar tudo e depois fazer PASTE.

13. Gravar o ficheiro com o nome 1IEP-ligand.pdb na pasta do projecto (seta vermelha). Não esquecer a terminação pdb. Deverá ter os átomos 4465 a 4500. Fechar o ficheiro. Nesta fase devemos ter a nossa pasta de projecto com os seguintes ficheiros.
 `
`
14. No ADT que abriram antes ir ao menu "File>Read Molecule" e escolher o ficheiro 1IEP-rec.pdb que escolheram antes.

14. Abriram o proteína e devem visualizar a proteína da seguinte forma.

Podem manipular a forma de visualizar a estrutura na secção branca indicada pelas setas laranja. Para movimentar a proteína neste visualizador pode utilizar os seguintes comandos:
- o botão do meio do rato para rodar a proteína.
- o botão direito do rato para movimentar a proteína.
- o botão do rato do lado direito mantendo a carregar no "Shift" para fazer zoom in/out
Experimente um pouco.
15. Agora vamos preparar correctamente a estrutura para o docking. Clicar no menu "Edit>Hydrogens>Add", escolher "Polar Only" (seta amarela) e clicar "OK".

Os hidrogénios polares deverão aparecer na imagem com cor azul clara.
Os hidrogénios polares não são normalmente representados nas estruturas de proteínas obtidas por cristalografia de raio-X, mas são necessárias para o docking pois podem ter influência no docking proteína-ligando.
16. Clicar no menu "Edit>Charges>Compute Gasteiger" e clicar "OK". Clicar no menu "Edit>Atoms>Assign AD4 type..." e clicar "OK".
Desta forma adicionamos as cargas a todos os átomos. No ficheiro original esta informação não é dada e é importante no docking pois as cargas afectam o docking proteína-ligando.
17. Gravar a proteína no formado pdbqt requerido para o docking. Clicar no menu "File>Save>Write PDBQT"(seta verde) e gravar na pasta do projecto a estrutura com o nome 1IEP-rec.pdbqt. Não esquece a terminação pdbqt.

18. Clicar no menu "Ligand>Input>Open" e abrir o ficheiro 1IEP-ligand.pdb que criou anteriormente (seta azul).

19. O ligando imatinib (STI-571) é aberto. Para visualizar o ligando como na figura abaixo clicar no rectângulo assinalado pela seta roxa de forma a esconder a proteína.

20. Clicar no menu "Ligand>Output>Save as PDBQT..." e gravar o ligando com o nome 1IEP-ligand.pdbqt (seta vermelha). Não esquecer a terminação pdbqt.

21. Nesta fase devemos ter a nossa pasta de projecto com os seguintes ficheiros.

(2) Realização do docking - AutoDock Vina
Agora vamos utilizar o software Vina para realizar o docking proteína-ligando.
22. Agora vamos definir um espaço Tridimensional de procura (Grid) dentro do qual o ligando vai tentar ligar à estrutura da proteína. Este espaço (Grid) deve abarcar a zona catalítica se existir, mas não deve ser grande demais pois neste caso o ligando pode não encontrar as zonas de ligação. Clicar no menu "Grid>Grid Box". Aparece a seguinte janela "Grid Options".

23. Clicar no menu "Center>Pick an Atom" e seleccionar um dos átomos centrais do ligando. Deverá observar um espaço tridimensional (Grid) na forma de cubo com lados vermelho, azul e verde.

24. Na janela "Grid options aumentar a opção "Spacing (angstrom)" para "1". Colocar todas as opções como na janela seguinte. Apontar os valores.

25. Para utilizar o programa Vina temos que criar um ficheiro de texto com o nome conf.txt e que deve apresentar as valores que apontou no passo anterior. Crie o ficheiro utilizando o WordPad e não se esqueça de o gravar na pasta do projecto. O ficheiro deve conter as seguintes linhas: o nome da proteína e do ligando que preparou antes (seta laranja), as coordenadas x y e Z do grid arrredondado ao valor inteiro mais próximo (seta amarela), o tamanho do grid (seta verde) e acrescente a linha apresentada a azul.
Atenção os nomes devem ser exactamente iguais aos nomes dos ficheiros que gravaram incluindo as terminações!
O ficheiro conf.txt deve ser gravado no formato de testo simples (.txt) e não Rich Text Format (.rtf)!

26. Abri a linha de comandos. Clicar no menu "Iniciar" pesquisar a frase "linha de comandos" e clicar no programa "Linha de comandos" que aparece nos resultados (setas amarelas). Em alternativa ir ao menu "Acessórios " e selecionar "Linha de Comandos". Vai aparecer a seguinte janela.

27. Dependendo do computador as linhas de comando que tem de escrever podem ser diferentes (setas brancas): (1) entrar na pen digitando "i:", (2) entrar na pasta projecto digitando "cd docking", (3) visualizar o conteúdo pa pasta digitando "dir", (4) correr o programa Vina digitando "vina.exe --config conf.txt --log 1IEP.log".

28. O processo de docking começa. O programa consegue detectar se tem dois processadores no computador e usa dos dois se existirem (1). Pode ver a evolução do ensaio até acabar (2). Com os parâmetros utilizados demorará à volta de 5-6 minutos.

29. No final do docking temos os resultados. o Vina encontra 3 modos de ligação sendo que o primeiro (com energia de ligação mais baixa - affinity de 13,0 Kcal/mol será o que nos interessa mais.

(3) Análise de resultados - ADT
30. Para visualizar a conformação de ligação na estrutura ir ao ADT e clicar em "File>Read Molecule", e e escolher o ficheiro "1IEP-ligand_out.pdbqt que o Vina criou na pasta do projecto. Alterar as configurações do ADT até conseguirem visualizar a a proteína e os ligandos no formato indicado na imagem abaixo.

Que informação é que podemos extrair da visualização dos da conformação de ligação?
31. Agora repetimos o processo para outros ligandos de interesse. Para o trabalho escolham pelo menos 2 destes 4 compostos: 393R, 460R, 560R ou 566R. Este compostos estão no e-learning e já estão no formato pdbqt. Para fazer docking com estes compostos basta trocar o nome do ligando no ficheiro conf.txt e correr outra vez o vina.exe.
Estes são os compostos que deram melhor actividade antitumoral, será que são potenciais inibidores das proteínas expressas pelo vosso gene?
32. Para o trabalho de pesquisa podem gravar imagens da proteína clicando no menu "File>Save>Save image as ..." e depois escolher uma pasta onde gravar a imagem. Esta imagem pode depois ser utilizada para ilustrar o trabalho.
Passo Extra!
1. Como visualizar a proteína com superfície electrostática. Onde está o ligando neste imagem?
