Aulas Teórico-Práticas de Bioquímica
|
| Bases de Dados |
| Alinhamento de sequências |
| OMIM |
| Visualização de proteínas 2 |
| Árvores Filogenéticas |
CLC Sequence Viewer
Neste tutorial vamos aprender a utilizar o programa CLC free workbench.
Existem muitos softwares ao dispor do investigador na área da Biologia Molecular Computacional. A maior parte deles são criados por investigadores cujo objectivo é normalmente o de criar ferramentas para lidar com problemas que ocorrem durante a sua pesquisa. Muitos destes investigadores acabam por disponibilizar os softwares sem custos, normalmente pela internet.
Apesar deste processo permitir uma rápida evolução dos softwares, também cria grande confusão na hora de decidir que ferramente utilizar. Além disso muitas vezes a facilidade de utilização não é o objectivo principal dos criadores destes programas. Assim o passo lógico seguinte é a junção de ferramentas em packs de software e uma maior atenção na facilidade de utilização destes packs.
O software que vamos utilizar é um dos produtos deste esforço e permite a manipulação de sequências de DNA, RNA e proteínas. Apesar de em termos de funções não ser o mais completo, é fácil de utilizar, graficamente mais apelativo (sempre importante), e principalmente é de acesso livre. De qualquer modo as ferramentas que vamos utilizar neste tutorial são comuns para outros programas e por isso não será complicado mudar para outro programa.
Outro pack mais completo é o Vector NTI Advance 11 lançado pela Invitrogen e é o mais utilizado em Biologia Molecular. Não foi o escolhido para esta tutorial porque não é tão fácil de utilizar e custa bastante dinheiro Mas as funções que vamos aprender neste tutorial podem ser transpostos para o Vector NTI Advance 11 com bastante facilidade.
Tutorial1. Antes de utilizar o programa é necessário abrir o site http://www.clcbio.com. Clicar nos links indicados pelas setas laranjas. De seguida clicar no link "Link to Download" indicado pela seta amarela que aparece ao lado do nome CLC Sequence Viewer.


2. Preencher o formulário indicando o email e o nome (setas vermelhas). Escolher o sistema operativo utilizado. Se não quiser receber email da empresa desmarcar no local "email notifications". Finalmente clicar no botão "Download" (seta vermelha).

3. Escolher o opção "Guardar Ficheiro" e escolher um locar para guardar o programa. Depois é só instalar o programa. Dizer que sim a tudo. O programa está instalado.

4. Se tudo correu bem na instalação deverá ter no desktop um link como este.

5. Faça duplo click no link para abrir o programa.
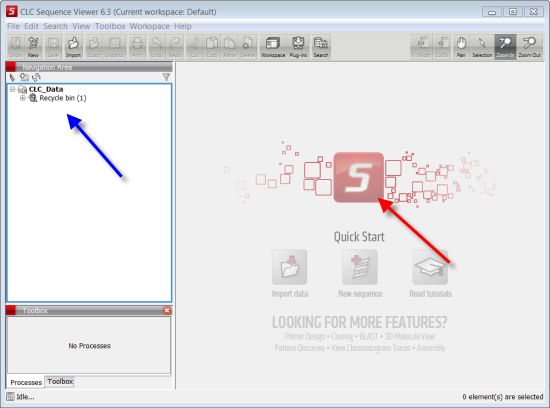
Ao abrir o programa pode observar a interface do utilizador com duas secções principais: a área de navegação indicada pela seta azul e a área de trabalho indicada pela seta vermelha.
A área de navegação é o local onde pode gravar e aceder a toda a informação de um determinado projecto incluindo a informação original e as análises realizadas pelo software.
A área de trabalho é onde a informação de um determinado ficheiro pode ser observada graficamente e onde normalmente se manipula e analisa a respectiva informação.
6. Quando abre o programa normalmente aparece na área de navegação um projecto "CLC_data". Neste tutorial vamos criar um projecto novo. Para isso clique em: "File>New>Folder" (seta verde). Escolha um nome para o projecto, por exemplo "PIGP" e clique "Enter".
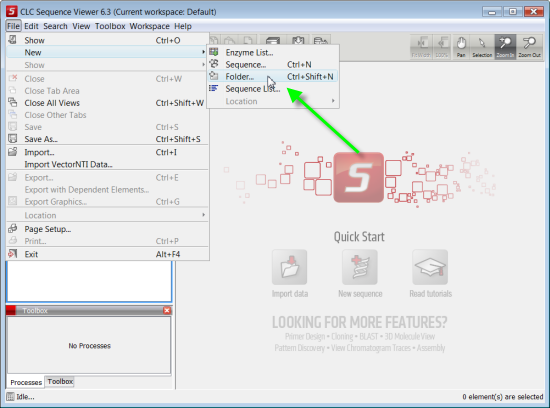
7. Dentro do projecto vamos criar outra pasta (folder) para sequências de DNA. Para isso clique com botão do lado direito do rato em cima do nome do projecto e escolha "New>Folder" (setas laranjas). Dê o nome DNA para a pasta.

8. Agora vamos importar uma sequência de nucleótidos. Para este módulo vamos utilizar a sequência de nucleótidos mRNA que gravamos no módulo de "Alinhamento de sequências". Para importar um ficheiro, selecionar a pasta "DNA" criada no passo anterior e clicar em "File>Import". Depois é só escolher o ficheiro de texto que gravaram anteriormente. O ficheiro foi gravado em formato FASTA que o programa reconhece. No meu caso gravei como "PIGP.txt".
9. Uma vez importada a sequência, ela aparece dentro da pasta DNA (seta azul). Para visualizar a sequência basta fazer duplo clique em cima do nome da sequência na área de navegação e ela aparece na área de trabalho (seta vermelha).
k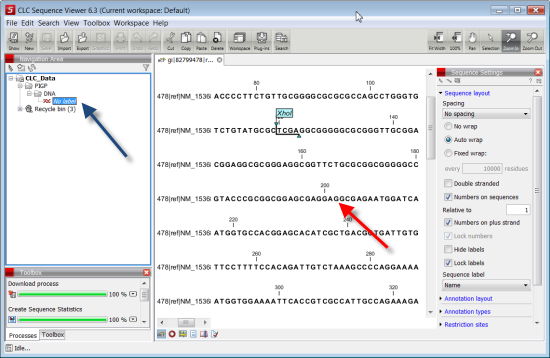
10. A sequência aparece sem nome (no label). Clicar com o botão do lado direito do rato no nome da sequência escolher Sequence Representation>Name (setas amarelas).

Como pode observar o nome aparece como gi|82799478|ref|NM_153681.2. A sequência utilizada neste tutorial tem 991 nucleótidos como se pode observar na área de trabalho e como visto na última aula.
Indicado pela seta verde pode ver quais os ficheiros que estão abertos ao mesmo tempo. Neste caso está aberta a sequência de DNA importada anteriormente.
A seta rosa indica a barra "View Preferences" onde pode alterar as configurações de visualização das sequências.
11. No local indicado pela seta rosa da figura anterior onde diz "Fix wrap " colocar 200 em vez que 10000.

Como se pode observar na figura a visualização é agora feita de 200 em 200 nucleótidos. Para alterar outros parâmetros da visualização utilize os botões indicados pelas setas da figura.
A seta azul indica dois botões, se clicar no botão "100%" pode observar todos os nucleótidos individualmente indicados pelas letras respectivas. Ao clicar em "Fit width" conseguimos observar esquematicamente toda a sequência dentro da janela de visualização.
A seta vermelha indica os botões de "Zoom in" e "Zoom out" com os quais pode ver a sequência em maior ou menor pormenor.
A seta verde indica o botão que nos permite deslocar a sequência de sítio.
Pode alterar outros parâmetros na barra "View preferences". Nesta altura do tutorial pode experimentar um pouco com os comandos alterando outros parâmetros na barra "View preferences".
12. Fazer segunda vez duplo clique no nome da sequência na área de navegação. Aparecem dois tabs com o nome da sequência. Num dos tabs clicar com o botão do lado direito do rato e escolher "View>Split Horizontally" com indicado na figura.

Apesar de ter aberto duas visualizações da mesma sequência na área de trabalho, ainda temos apenas um sequência na área de navegação. Estamos apenas a visualizar a mesma sequência de duas formas diferentes.
13. Alterar as duas visualizações de forma a ficarem como na figura indicada abaixo.
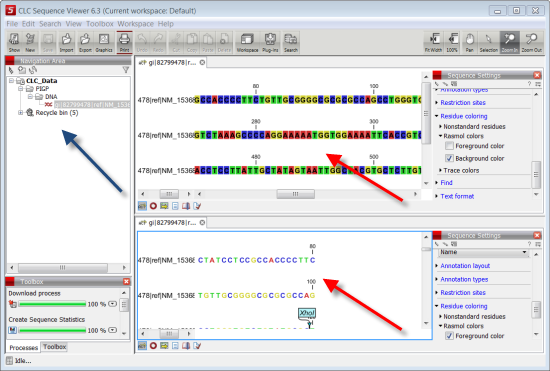
Com as duas visualizações podemos trabalhar numa parte da sequência sem perder noção da sequência completa. Como foi referido anteriormente temos duas visualizações diferentes (setas vermelhas) para a mesma sequência (seta azul).
14. Clicar no botão "Selection" indicado na figura seguinte pela seta verde. Selecionar os nucleótidos indicados pela seta amarela.
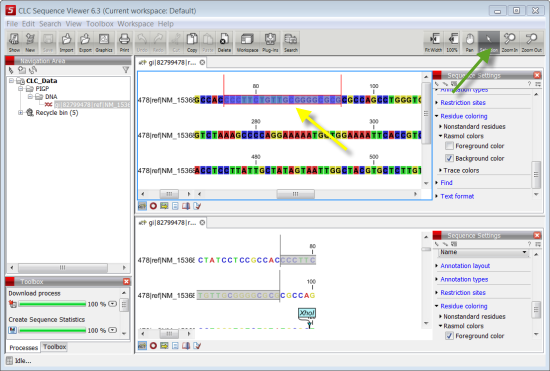
Como se pode observar na figura, ao seleccionar uma parte da sequência numa das janelas de visualização, também é selecionada na outra janela de visualização a mesma parte da sequência. Desta forma podemos estar a observar uma sequência em detalhe sem perder a noção geral da sequência.
15. Agora que já utilizamos as ferramentas básicas para visualização de sequências, vamos pesquisar sequências na NCBI directamente do programa. Para isso é necessário estar ligado à internet. Preencher o formulário que aparece com a palavra chave "PIGP" indicadas na figura (seta azul). Clicar em "Start Search" (seta verde). Os resultados aparecem abaixo (seta vermelha).

O programa vai pesquisar directamente na NCBI por sequências em que a expressão PIGP esteja presente. Podemos também procurar sequências de proteínas, para isso basta escolher"Proteins" em vez de "Nucleotide". Dos resultados encontrados, procurar a sequência que importamos anteriormente.
16. Seleccionar sequências de mRNA de: Homo Sapiens (2), Mus Musculus (6), Bos Taurus (1), Rattus Norvegicus (1) e Nasonia vitripennis (1), Oncorhynchus mykiss (1), Salmo Salar (1). No final devemos ficar com 11 sequências. Para colocar sequências no directorio DNA que criaram anteriormente basta fazer "drag-and-drop" Destas sequências. Para seleccionar várias sequências de cada vez manter a tecla "CTRL" pressionada enquanto selecciona as sequências. Deverá ficar com as sequências na pasta DNA (seta roxa).

Os resultados apresentados serão os mesmos do que se procurar directamente na base de dados da NCBI, como foi visto no módulo de bases de dados. Utilizar o software para pesquisar facilita bastante o trabalho embora os opções de pesquisa sejam mais limitadas.
Arrastar as sequências seleccionadas para a pasta DNA. Os nomes das sequências deverão aparecer sob a pasta DNA (seta rosa) Ao arrastar as sequências para a pasta estas são automaticamente gravadas, até serem arrastadas elas não estão gravadas, são apenas resultados de uma pesquisa.
17. Para ver o nome das espécies (em latim ou nome comum) a que pertencem as sequências clicar com o botão do lado direito na directoria DNA, clicar em "Sequence Representation>Latin Name (Accession)" ou "Sequence Representation>Common Name (Accession)"

18. Agora vamos alinhar as sequências escolhidas. Clicar no menu "ToolBox>Alignment and Trees> Create Alignment". Selecionar as sequências de Homo Sapiens (2) e Mus Musculus (6) que foram gravadas na subpasta "DNA" (mantenha-se a carregar em CTRL para selecionar mais do que uma sequência de cada vez). Logo que tiver colocadas as 8 sequências na janela da direita, clicar em "Next" e depois "Finish". O software começa a fazer o alinhamento que demora 1 ou 2 minutos (quantas mais sequências mais tempo demora). Podemos ver a percentagem que falta no canto inferior esquerdo.


Repare que o que vamos agora fazer é o alinhamento das sequências selecionadas. No módulo alinhamento de sequências fomos tentar alinhar uma sequência com todas as outras para verificar quais as mais homólogas da nossa sequência. Neste caso vamos alinhar apenas as sequências que escolhemos.
19. No final do processamento deverá obter um resultado como o indicado na figura. Para visualizar o resultado completo clicar em "Fit witdh " (seta amarela). Depois clicar em "100 % " (seta azul).

20. Em "Fixed Wrap", colocar 2000 e clicar em "Fit width".

Agora pode observar as sequências no seu comprimento completo e devidamente alinhadas. Que conclusões pode tirar sobre a naturesa das sequências deste alinhamento?
21. Fazer "Drag-and-Drop" do alinhamento para a directoria do DNA. Desta forma está a gravar o alinhamento na mesma pasta das sequências (seta vermelha).
 $
$
Repare que o símbolo do resultado é diferente dos ficheiros das sequências de nucleótidos.
22. Clicar no menu "Toolbox>Alignment and Trees>Create Tree". Escolher o alinhamento obtido nos passos anteriores. Clicar sempre em "Next" e no fim em "Finish".

23. Deverá obter um resultado como o indicado na figura seguinte.

24. Alterar as "Tree Settings" de forma a visualizar a árvore filogenética da seguinte forma.

Que conclusões pode obter desta "árvore filogenética"?
25. Fazer outro alinhamento de sequências com as seguintes sequências: Homo Sapiens (NM_153681), Mus musculus (NM_001159617), Bos Taurus (NM_001077022), Rattus Norvegicus (NM_001099758), Nasonia vitripennis (NM_001161589), Salmo salar (NM_001141884) and Oncorhynchus mykiss (BT_074395)..
Que conclusões pode obter desta segunda "árvore filogenética"?