Aulas Teórico-Práticas de Bioquímica
|
| Bases de Dados |
| Alinhamento de sequências |
| OMIM |
| Visualização de proteínas 2 |
| Árvores Filogenéticas |
Visualização de proteínas
Neste pequeno módulo vamos aprender a utilizar um software de visualização de estruturas 3D de proteínas. Ao longo do módulo vão verificar que a visualização destas estruturas permite compreender melhor os vários níveis organizacionais e a função das proteínas.
Primeiro que tudo é necessário saber onde obter as estruturas na internet. Para isso vamos utilizar a base de dados de estruturas do NCBI (National Center for Biotechnology Information).
Cada passo deste módulo vem acompanhada de uma imagem representativa. Depois da imagem temos a vermelho explicações suplementares sobre esse passo.
A primeira proteína que vamos utilizar como exemplo é a hexocinase. Então vamos começar...
1. Começar por clicar com o botão do rato do lado direito no site "entrez"da NCBI, http://www.ncbi.nlm.nih.gov/gquery/gquery.fcgi?itool=toolbar e escolher "abrir num novo separador" (ou "abrir numa nova janela"). Desta forma ficamos com o site do NCBI num separador ou janela e as instruções deste módulo noutro separador ou janela. Agora é só ir passando de uma janela para a outra e seguir as instruções. Escrever no campo à direita de "Search across databases" o nome da enzima "hexokinase" (seta vermelha).
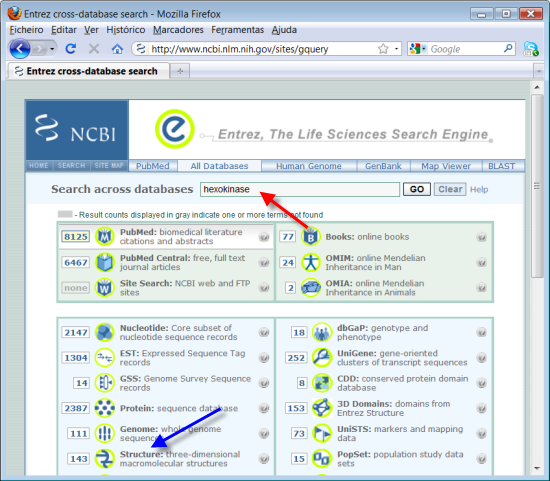
Como estamos a utilizar bases de dados que utilizam o Inglês como língua base, temos que realizar as pesquisas em Inglês, neste caso escrevemos hexokinase e não hexocinase.
Podemos observar a ocorrência de muitos resultados nas várias bases de dados disponibilizadas pela NCBI. Neste site procuramos muitas bases de dados ao mesmo tempo, incluíndo artigos científicos (Pubmed); sequências de nucleótidos (Nucleotide) e, a que nos interessa para esta aula, estruturas 3D de proteínas (seta azul).
2. Reduzir os resultados da pesquisa da enzima. Escrever no campo à direita de "Search across databases"o seguinte "hexokinase[Title]" (seta laranja).
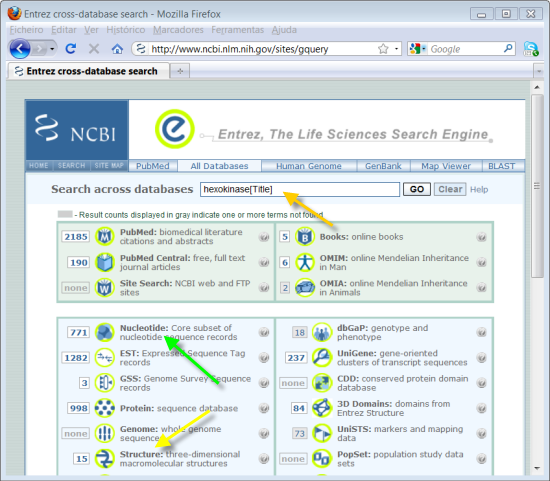
Ao colocar a palavra Title entre parêntices rectos, estamos a especificar que a pesquisa só deverá ser feita no título das entradas das bases de dados. Para visualizar estruturas de proteínas, interessa-nos a base de dados de estruturas 3D de proteínas, em inglês "Structure" (seta amarela). Podemos observar que temos apenas 15 resultados nesta bases de dados, quando comparados por exemplo com a base de dados de sequências de nucleótidos ("Core Nucleotide") - seta verde com 771. Este número limitado de estruturas é natural, uma vez que as técnicas utilizadas para a determinação de estruturas 3D de proteínas (Cristalografia de raio-X e NMR) são complexas e o volume de estruturas que conseguem gerar ainda é limitado.
3. Clicar na base de dados "Structure".

Podemos agora observar a base de dados "Structure" da NCBI, com os 15 resultados da hexocinase. Cada um dos resultados representa uma estrutura ou parte de uma estrutura 3D de uma proteína, obtida experimentalmente. Das 15 estruturas 3D de proteínas, podemos ver que: 11 são estruturas de organismos eucariotas, 4 são de bactérias; 14 foram obtidas com um ligando (substrato, coenzima ou ambos) e que as 15 estruturas foram obtidas por cristalografia de raio-X (X-ray) não sendo nenhuma obtida por NMR.
4. Seleccionar o resultado com o nome de "1QHA".
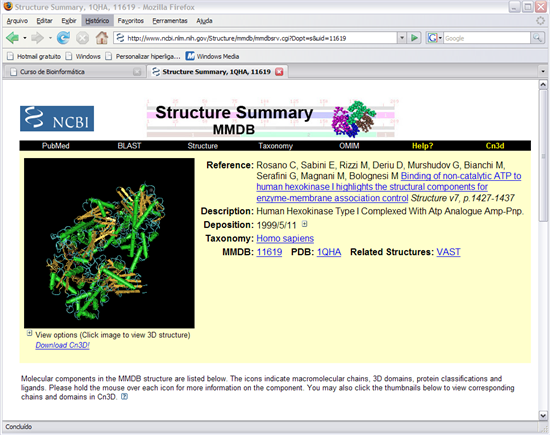
Escolhemos este resultado porque é a estrutura 3D da hexocinase humana. Para além disso apresenta características interessantes de visualização: tem duas subunidades e está ligada a um análogo da sua coenzima. A coenzima da hexocinase é o ATP, o análogo do ATP utilizado é o AMP-PnP. O AMP-PnP é utilizado pois este mantêm-se ligado permanentemente o que permite estudar a conformação da hexokinase com o coenzima ligada.
5. Clicar na imagem da proteína. Pode demorar alguns segundos a aparecer a estrutura. Se está a utilizar o seu próprio computador, deve fazer o download do programa, clicando em "Download Cn3D!" e realizar a instalação, antes de clicar na imagem da proteína.
Ao clicar na estrutura, o programa Cn3D é automaticamente aberto e a estrutura da proteína em questão pode ser imediatamente visualizada. Pela estrutura pode observar-se que a proteína tem estrutura quaternária, uma vez que apresenta duas subunidades homólogas. Apesar da estrutura desta proteína ter sido obtida por Cristalografia de Raio-X, o que estamos a observar é apenas um modelo. Temos apenas uma representação simplificada da proteína.
Para facilitar a visualização, as estruturas secundárias são apresentadas utilizando formas geométricas. Nesta caso os cilindros verdes representam hélices-alfa e as setas castanhas representam folhas-beta. Os loops entre as estruturas secundárias estão representadas a azul.
6. Na outra janela que foi aberta seleccionar mais ou menos 30 aminoácidos (as letras representadas em sequência).
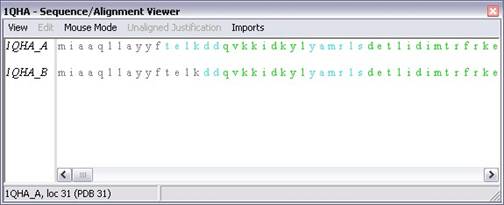
Na segunda janela aberta está representada a sequência de aminoácidos de ambas as subunidades (1QHA_A e 1QHA_B), na nomenclatura de 1 letra por cada 1 aminoácido. As cores em que estão representados os aminoácidos, correspondem à estrutura secundária de que fazem parte na estrutura da proteína. Se seleccionar alguns aminoácidos, estes aparecem selecionados na estrutura 3D a amarelo. Pode fazer esta selecção também para apenas um aminoácido, mas nesse caso vai ser mais difícil encontrá-lo.
7. Posicionar a proteína de forma a visualizar o AMP-PNP (o análogo da coenzima ATP), a glucose (o Substrato da enzima) e a glucose-6-fosfato (o Produto da enzima).
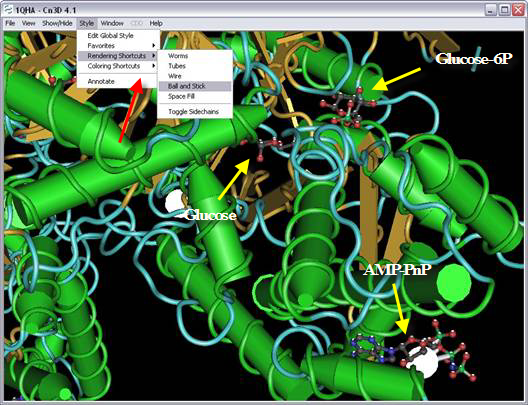
Para rodar a molécula, apenas tem de utilizar o rato mantendo-se a carregar no botão esquerdo do rato. Para fazer zoom na estrutura, mantenha a tecla Ctrl pressionada enquanto utiliza o rato. Para posicionar a estrutura sem a fazer rodar, mantenha a tecla para maiúsculas pressionada e utilize o rato.
Nesta configuração de visualização, apenas os ligandos da enzima são apresentados com os seus átomos constituintes. Cada cor representa um tipo de átomo:
Cinzento-Carbono
Vermelho-Oxigénio
Azul-Nitrogénio
Verde-Fósforo
Amarelo-Enxofre
Branco-Hidrogénio
Na imagem, pode ver-se os constituintes do ATP (na forma alterada AMP-PnP): a adenina com os seus anéis de carbonos e nitrogénios, a ribose com o seu anél de 4 carbono e um oxigénio e os 3 grupos fosfatos característicos. Os grupos fosfato são facilmente observáveis pelos seus átomos de fósforo representados a verde. O nitrogénio (azul) que se encontra entre os dois grupos fosfato, é a única difereça entre o AMP-PnP e o ATP (que tem um fósforo em vez do nitrogénio).
Muitas vezes os hidrogénios não são representados para facilitar a visualização. É o que acontece nesta estrutura. Na imagem podemos ver também onde se liga o Substrato (glucose) e o Produto (glucose-6-fosfato). A diferença entre elas encontra-se no grupo fosfato que se encontra ligado ao carbono 6 da glucose. Novamente, é fácil observar a diferença pelo átomo de fósforo (verde) do grupo fosfato.
8. Clicar no menu "Style", escolher a opção "Rendering Shortcuts", e depois a opção "Ball and Sticks" (seta vermelha).
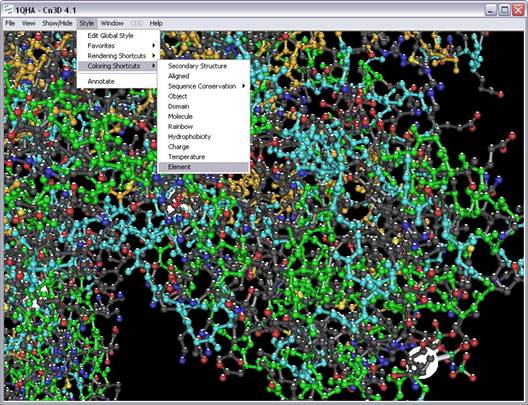
Com esta opção pode visualizar todos os átomos constituintes (excepto os hidrogénios) de todos os aminoácidos da proteína. Existem outras formas de visualização da estrutura 3D da proteína, experimente com outras formas. Esta forma de visualizar a estrutura das proteínas já é mais próxima da realidade. No entanto as proteínas não são estruturas estáticas, estão em constante movimento. A parte do movimento ainda é complicado de estudar e representar. Ddevemos ter sempre a noção que estas representações estáticas das proteínas são como "fotografias" tiradas a algo em constante movimento.
9. Clicar no menu "Style", escolher a opção "Coloring Shortcuts", e depois a opção "Elements".

Com esta opção vai poder observar todos os átomos de acordo com as cores indicadas no passo 11. Repare nos átomos de enxofre que se encontram representados a amarelo e que indicam normalmente uma ponte dissulfeto (ou de enxofre). Também podemos observar os aminoácidos de acordo com várias das suas características como a carga ou a hidrofobicidade. Experimente algumas das opções.
10. Clicar no menu "File", escolher a opção "Export PNG ".
Se pretende gravar alguma imagem estática da molécula que ache interessante ou que queira utilizar para um trabalho, basta exportar a molécula no formato de imagem PNG. Depois é só inserir a imagem no documento que pretender, por exemplo um documento word.
11. Manter os settings como estão e clicar OK.
12. Escolher uma pasta para gravar o ficheiro e dar um nome ao ficheiro.
Pronto. Agora pode utilizar a imagem para,por exemplo, ilustrar um trabalho. Não esquecendo claro, de referenciar a fonte bibliografica da imagem!
13. Clicar no menu "View", escolher a opção "Animation", e depois a opção "Spin" (ou clicar na tecla n).
Com esta opção pode colocar a proteína a rodar. Na opção "Set Delay" pode mesmo configurar a velocidade com que a molécula roda. Para parar de rodar a proteína basta escolher a opção "Stop (ou basta carregar na tecla s).
Se quiser visualizar a proteína sem estar ligado à net basta gravar a molécula no formato"asn".
O programa CN3D tem muitas outras funções mas com este módulo ja aprenderam o suficiente para explorarem sozinhos o software. Existem softwares melhores, mas mais complexos, se estiverem interessados podem tentar estes dois de acesso livre:
PYMOL (http://pymol.sourceforge.net)
DEEPVIEW - SWISS PDB (http://www.expasy.org/spdbv/)
Vamos ainda estudar outra proteína mais conhecida, a hemoglobina.
15. Voltar ao site da NCBI,
http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?CMD=search&DB=structure. Escolher abrir o site noutra página.
16. Pesquisar "hemoglobin deoxy homo sapiens" (seta vermelha). Clicar Go.
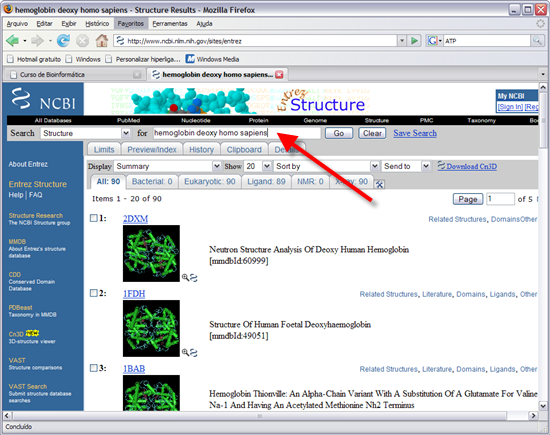
Temos 93 estruturas 3D da proteína hemoglobina. O termo "deoxy" significa que estamos a pesquisar estrutura da hemoglobina, sem o Oxigénio ligado. Como já devem saber a função da hemoglobina é o transporte de Oxigénio dos pulmões para os tecidos do organismos onde este é necessário. Com o termo "Homo Sapiens" especificamos que estamos à procura de estruturas de hemoglobina da espécie humana.
17. Procurar e clicar o resultado "1XZ2" (deverá estar na terceira página de resultados).
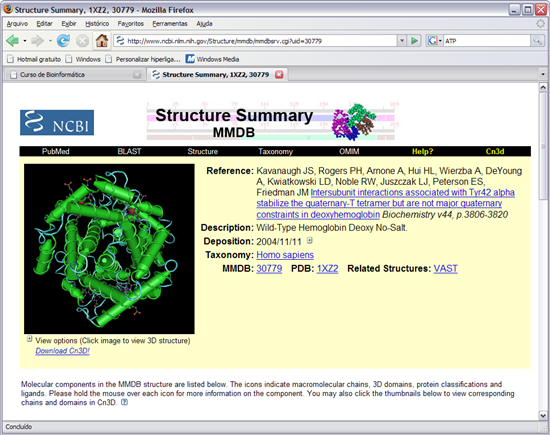
Como podem ver, a estrutura da hemoglobina humana tem 4 subunidades (Chain A, B, C, D) cada uma liga e transporta uma molécula de Oxigénio.
18. Vamos então observar uma estrutura da hemoglobina. Clicar na imagem da estrutura.
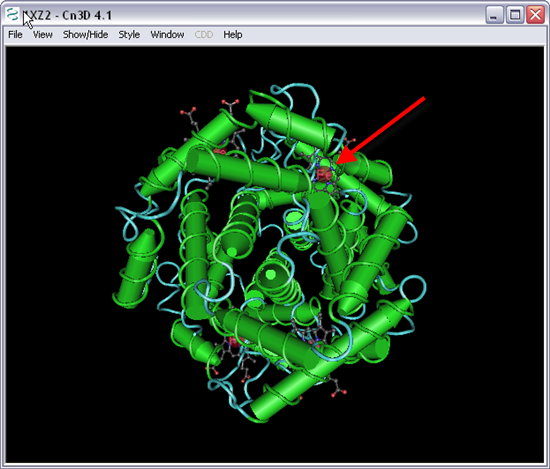
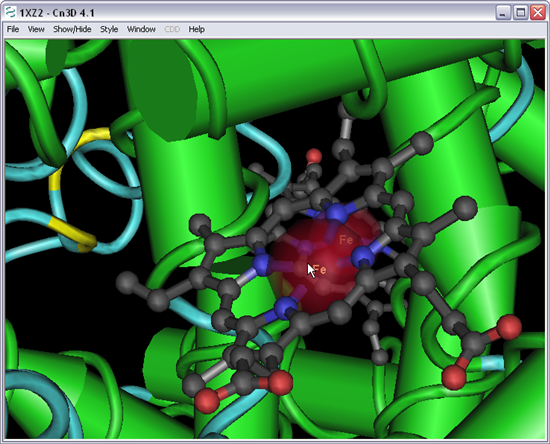
Cada subunidade da hemoglobina tem 1 grupo heme com um ião Ferro (Fe2+) no centro de cada grupo heme (seta vermelha).
19. Posicionar a proteína de forma a que seja possível ver de perto um dos grupos heme e o ião Fe2+.
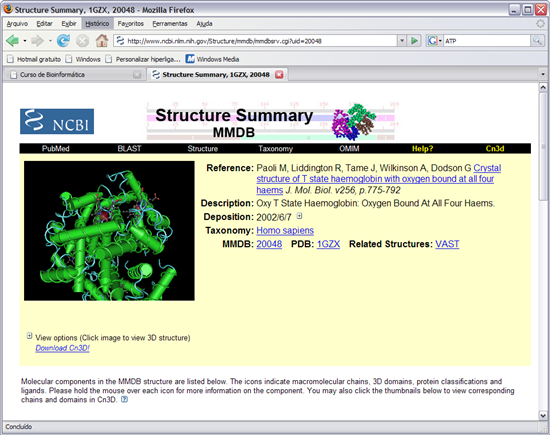
20. Vamos ainda procurar uma outra estrutura da hemoglobina. Voltar à página de pesquisa. Pesquisar com as palavras "hemoglobin oxy homo sapiens" (seta vermelha).
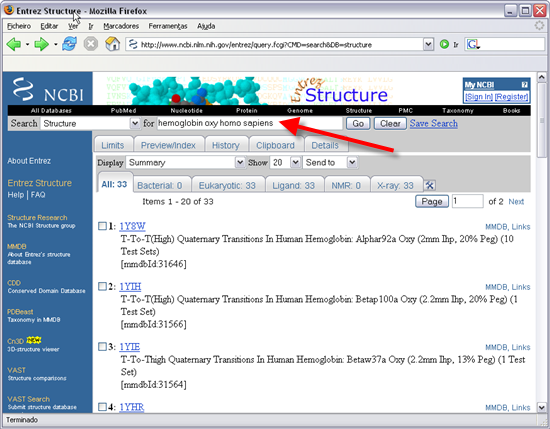
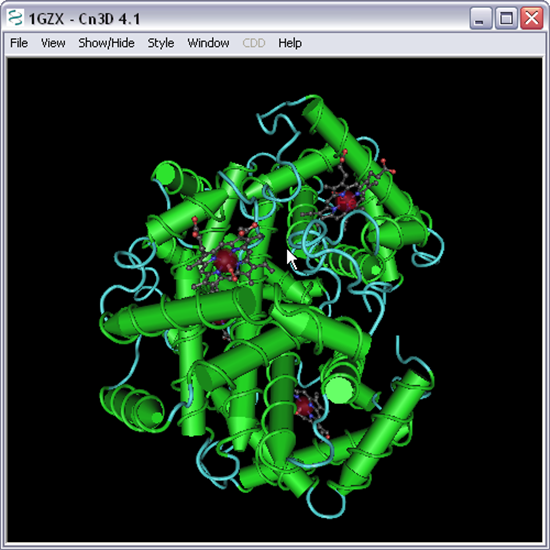
21. Procurar e clicar o resultado "1GZX" (deverá estar na segunda página de resultados).

22.Clicar na estrutura da proteína.
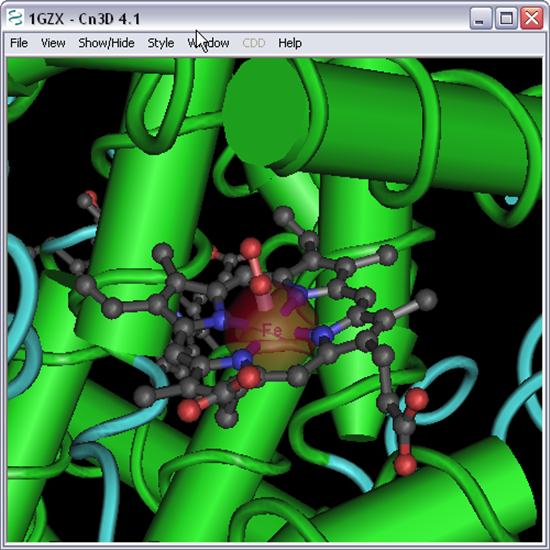
23. Manipular a hemoglobina de forma a observar em pormenor o grupo heme. Qual a diferença entre as duas estruturas de hemoglobina observadas?
PDB - Protein DataBank
24. Abrir uma página com o seguinte endereço:
(http://www.rcsb.org/pdb/Welcome.do).

Este é o site da PDB (Protein DataBanK) e é um dos recursos mais importantes para quem pretende informação sobre a estrutura 3D de proteínas.
25. Escrever no campo à direita de "Search" a enzima "hexokinase".
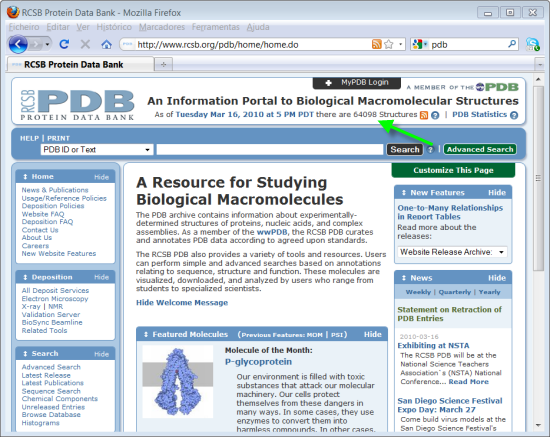
Podemos observar que os resultados são aproximadamente os mesmos, que na pesquisa na base de dados "Structure" da NCBI com algumas diferenças provocadas pelos métodos de pesquisa. Isto é de esperar pois ambas as bases de dados de estruturas estão sincronizadas.
Repare que na parte de cima podemos ver o número de estruturas actualmente no PDB, cerca de 64098. Este número é actualizado semanalmente.
26. Selecionar novamente o resultado 1QHA.

O site PDB tem bastante mais informação para cada estrutura. Do lado direito podemos ver várias opções: download da proteína, visualização de proteínas ("display molecule") bem como informação mais específica sobre a estrutura incluíndo parâmetros de qualidade ("Resolution", "R-value", "R-free"), etc.
27. Selecionar o link "EC" que se encontra mais abaixo numa pequena bola com as letras EC (seta vermelha).
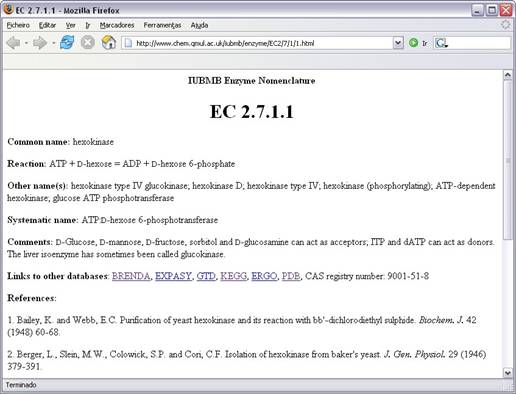
Nesta página podemos ver qual a nomenclatura sistemática (IUBMB Enzyme Nomenclature) da enzima que é composta por 4 números separados por pontos (neste caso EC 2.7.1.1). A partir desta página podemos obter mais informações sobre a enzima. Mais importante temos links para várias bases de dados com informação diversa sobre a enzima em estudo.
28. Selecionar o primeiro link "BRENDA".
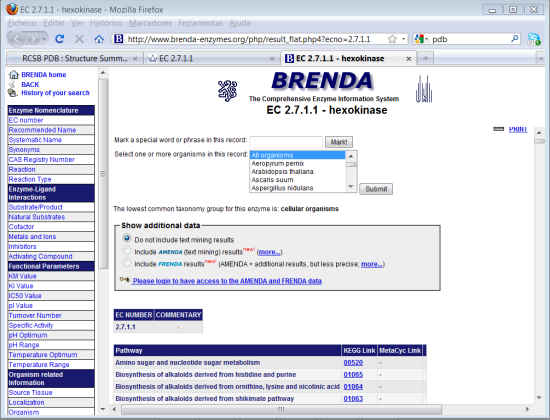
Este site tem informação muito detalhada sobre cada enzima. Atenção que apenas pode aceder a este site dentro da rede da escola, e necessita de introduzir o username e password que utiliza na escola. Do lado esquerdo temos "links" para várias características da enzima: informação sobre a ligação enzima-ligando (substratos, cofactores, inibidores, activadores) até informação sobre parêmetros funcionais como Km, Ki, Turnover, actividade específica, pH e temperatura óptima (Importante para a aula prática de Cinética Enzimática). Pesquise alguns dos "links".
29. Voltar ao site da nomenclatura (IUBMB Enzyme Nomenclature). Clicar em "KEGG". 
Neste site podemos obsevar mapas sobre vários aspectos da biologia molecular, relacionadas com a enzima em estudo. No nosso caso apresenta, para além de alguma informação diversa sobre a enzima, os mapas das vias metabólicas em que esta enzima está envolvida.
30. Clicar no link "map00010" da Glycolysis/Gluconeogenesis.
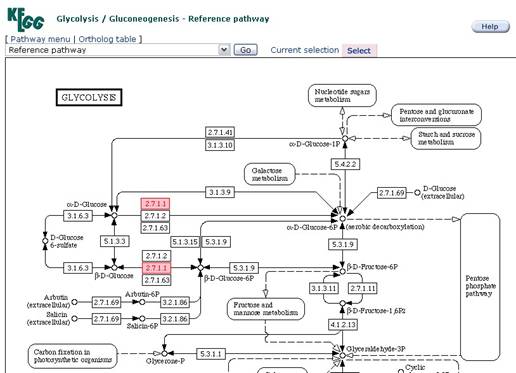
Podemos observar o posicionamento da enzima na via metabólica (sombreado a cor de rosa). Pesquise nos mapas das outras vias metabólicas em que a enzima está envolvida.
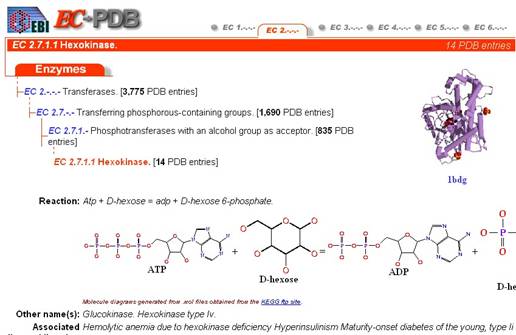
Este site (EC-PDB) é subsidiário do site do PDB e apresenta todas as enzimas conhecidas de acordo com a sua nomenclatura sistemática (IUBMB Enzyme Nomenclature). Podemos observar que a hexocinase é: da classe das transferases (2), da subclasse das transferases que transferem grupos fosfato (2.7), da sub-subclasse das transferases que transferem grupos fosfato e com um grupo alcool como grupo aceitados (2.7.1). O último número corresponde à ordem de descoberta da enzima EC 2.7.1.1 - no caso hexocinase foi a primeira a ser descoberta deste grupo).